Développement du modèle RBA pour prédire la réponse des cellules végétales dans des conditions environnementales complexes
Dans le cadre du métaprogramme INRAE DIGIT-BIO (Biologie numérique pour explorer et prédire le vivant), le projet exploratoire PlantRBA (2021 - 2023) visait à développer, calibrer et valider expérimentalement un modèle mathématique prédisant le comportement métabolique de la plante Arabidopsis thaliana sous contraintes abiotiques (limitation de la disponibilité en eau et/ou en azote). Ce modèle se base sur la répartition parcimonieuse des ressources entre les différentes fonctions biologiques de la plante et réconcilie ainsi les échelles les plus fines (les gènes, les métabolites) au phénotype.
Prédire la réponse des cellules végétales dans des conditions environnementales complexes représente un défi en biologie végétale. Les équipes INRAE MaIAGE, PHYGERM, SATURNE et VAST ont développé en collaboration un modèle d'allocation des ressources à l'échelle cellulaire et moléculaire pour la cellule photosynthétique de la feuille d'A. thaliana, basé sur une modélisation contrainte de type ‘Resource Balance Analysis’ (RBA). Le modèle RBA contient le réseau métabolique et les principaux processus macromoléculaires localisés dans les compartiments cellulaires impliqués dans la croissance et la survie de la cellule végétale. Le modèle simulé pour des conditions environnementales variables telles que la température, l'irradiance, la pression partielle de CO2 et O2, a permis de comparer les prédictions RBA avec les distributions de ressources connues et les traits phénotypiques quantitatifs, tels que le taux de croissance relatif, le rapport C/N, et enfin les caractéristiques empiriques de la fixation du CO2 données par le modèle bien établi de Farquhar. En comparaison avec d'autres méthodes de modélisation contrainte standard, telles que les modèles ‘Flux Balance Analysis’, le modèle RBA fait des prédictions quantitatives précises sans avoir besoin de contraintes empiriques. Ainsi, les résultats obtenus montrent que le RBA améliore significativement la prédiction autonome des phénotypes cellulaires végétaux dans des conditions environnementales complexes, et fournit des liens mécanistiques entre le génotype et le phénotype de la cellule végétale. Ils ont fait l’objet d’une publication scientifique dans Metabolic Engineering.
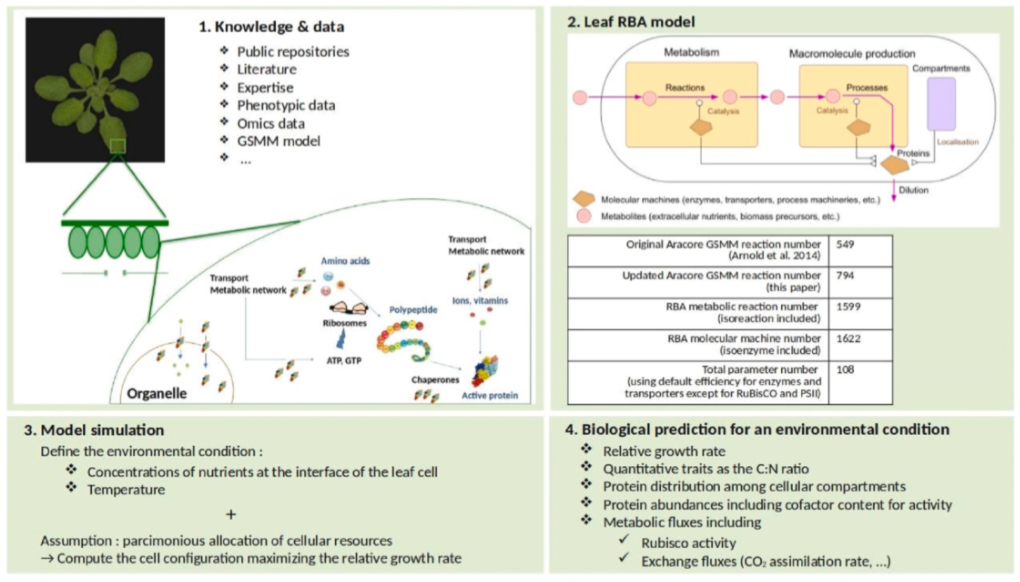
Principe de génération et simulation du modèle RBA (Figure de Goelzer et al., Metabolic Engineering, 2024)
Les recherches se poursuivent actuellement grâce au projet ANR PRC de recherche collaborative ModLsys (2023-2028), pour établir -et partager avec la communauté- un cadre de modélisation multi-échelle pour les systèmes vivants.
Une recherche développée à l’Institut Jean-Pierre Bourgin - Sciences du Végétal
Retour
Principe de génération et simulation du modèle RBA (Figure de Goelzer et al., Metabolic Engineering, 2024)
Zoom
Référence :
A Goelzer, L Rajjou, F Chardon, O Loudet, V Fromion. (2024). Resource allocation modeling for autonomous prediction of plant cell phenotypes. Metabolic Engineering, doi : https://doi.org/10.1016/j.ymben.2024.03.009
Contact :
Olivier Loudet, contact
Équipes IJPB :
> "Physiologie de la germination" PHYGERM
> "Senescence, Autophagie, Recyclage Nutritionnel et Efficacité d'Utilisation de l'Azote" SATURNE
> "Variabilité et Tolérance aux Stress Abiotiques" VAST
Équipe collaboratrice :
"Mathématiques et Informatique Appliquées du Génome à l'Environnement" MaIAGE