L’équipe SAS de l’IJPB ouvre la voie vers la compréhension des mécanismes allélopathiques
Face au défi de la transition agroécologique et de la réduction massive de l’usage des pesticides, exploiter les propriétés ‘allélopathiques’ des cultures apparaît comme une approche pertinente pour favoriser la régulation biologique des adventices. L’allélopathie correspond à un mécanisme par lequel une plante, via la libération de métabolites spécialisés, affecte positivement ou négativement, la croissance des plantes voisines indépendamment de relations trophiques. Cependant, favoriser la régulation des adventices par la mobilisation des propriétés allélopathiques demeure un processus complexe à mettre en place. En effet, les effets de l’allélopathie (en soi) sont difficiles à qualifier et à quantifier sur les plantes cibles eu égard à la complexité des interactions entre plantes comme la compétition pour les ressources. Par ailleurs, les mécanismes moléculaires impliqués sont très mal connus, ainsi que le contrôle génétique et environnemental des effets allélopathiques. 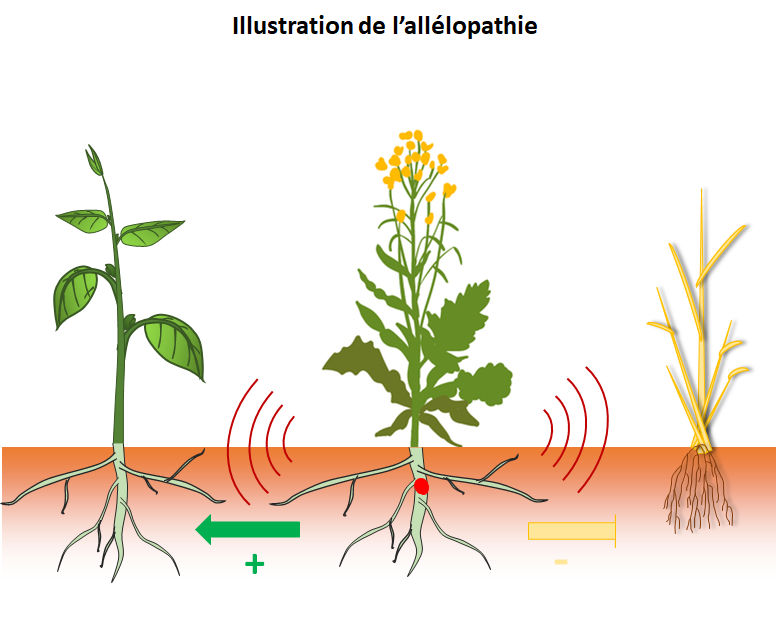
L’équipe SAS (Signalisation Allélopathique et Strigolactone) de l’IJPB s’est donnée l’objectif d’identifier les gènes et les signaux chimiques de la rhizosphère impliqués dans les interactions plante-plante et de comprendre comment ces signaux peuvent affecter le développement d’autres plantes pour ainsi contribuer à l’essor des pratiques agroécologiques. Pour cela, l’équipe développe 2 axes de recherches : le premier repose sur une approche non-ciblée de génétique d’association et de métabolomique des exsudats racinaires, le second vise à caractériser une famille de récepteurs de composés allélopathiques, les protéines D14/KAI2.
Des gènes d’intérêt potentiellement impliqués dans l’allélopathie intra-spécifique
Grâce au financement du projet ALLELO (2019-2021) par l’Institut Carnot Plant2Pro, l’équipe SAS, en collaboration avec l’IGEPP de Rennes et le LIPME de Toulouse, a mis en place un dispositif innovant de phénotypage des propriétés allopathiques en conditions contrôlées, appelées « Plant Soil Feedback (PSF) » permettant de s’affranchir des effets de la compétition. Des approches de génétique d'association sur la plante modèle Arabidopsis thaliana grâce à ce dispositif, et a l’utilisation du Phenoscope de l’Observatoire du végétal de IJPB, une quinzaine de gènes candidats potentiellement impliqués dans l’allélopathie intra-spécifique, ont été cartographiés. Deux d’entre eux sont impliqués dans le métabolisme des glucosinolates, une grande famille d’hétérosides soufrés, propres à l’ordre des Brassicales. Ces métabolites sont des acteurs de la défense des plantes et sont connus pour être exsudés/libérés par les racines chez arabidospsis et pouvant avoir des propriétés allélopathiques. Cependant, leur rôle doit encore être validé et le transfert à des espèces cultivées, comme le colza (espèce d’intérêt agronomique majeur) ou la cameline, est nécessaire dans une perspective d’application agronomique. Afin de permettre la validation de ces gènes candidats, une thèse financée à 100% par le département BAP d’INRAE débutera en octobre 2022. Elle a pour objectif de valider l’implication des glucosinolates dans l’allélopathie, ainsi que d’identifier la nature exacte des glucosinolates ou dérivés responsables de ces effets. Les travaux de l’équipe viennent d’être soutenus par le consortium PlantAlliance avec le financement du projet ALLOGA à l’interface entre génétique et agronomie. Ce projet conduit en collaboration avec l’GEPP de Rennes et l’unité Agroécologie de Dijon, a pour objectif d’adapter au colza les systèmes expérimentaux de PSF mis au point sur arabidopsis afin d’évaluer les propriétés allélopathiques d’un large panel de variétés de colza. Les tests seront aussi appliqués en inter-spécifique entre Arabidopsis et différentes espèces de plantes adventices rencontrées dans des populations d’Arabidopsis. Les connaissances obtenues permettront d’intégrer l’allélopathie dans un modèle mécanistique et ainsi quantifier les effets des systèmes de culture sur la dynamique d'une flore adventice. Ces données pourront être utilisées pour évaluer les propriétés allélopathiques de colza en culture.
Des récepteurs de signaux allélopathiques « aux multiples facettes »
Le second axe du projet de recherche de l’équipe SAS repose sur les connaissances acquises sur les protéines D14/KAI2 qui sont les récepteurs des strigolactones. Les travaux de l’équipe sur différentes espèces comme Pisum sativum (pois), Arabidopsis thaliana, Physcomitrium patens (mousse) ou encore les plantes parasites Phelipanche ramosa, et Orobanche cumana nous ont permis de démontrer que ces protéines étaient impliquées dans la perception d’autres composés allélopathiques. Dernièrement, nous avons démontré que les protéines PpKAI2 (au nombre de 11) de la plante non-vasculaire, P. patens, étaient impliquées dans le développement de cette mousse et la perception de signaux allélopathiques (1,2). Afin d’identifier le ou les signaux allélopathiques perçus par ces protéines, l’équipe SAS en collaboration avec l’Institut de Chimie des Substances Naturelles a récemment développé des sondes et des molécules qui permettent de mieux comprendre leur fonction et d’élaborer des stratégies d’identification des ligands (3,4). L’obtention de la structure cristallographique des deux protéines KAI2 de pois, en collaboration avec l’université UC Davies, a permis de mettre en évidence un évènement de duplication des protéines KAI2 chez les légumineuses qui leur confère la capacité à percevoir une plus grande diversité de signaux rhizosphériques (5). 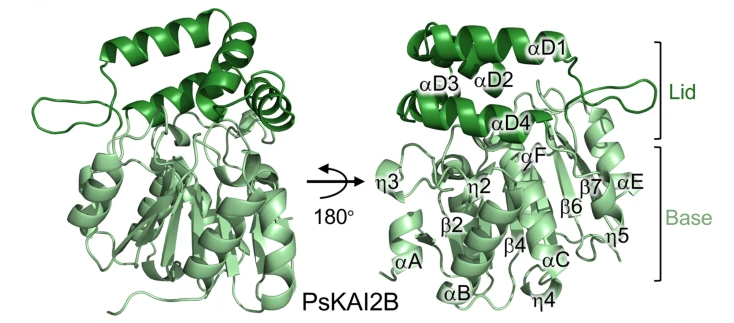
Vue d'ensemble de la structure cristalline de PsKAI2B (pois). Les domaines du couvercle et de la base sont colorés en vert foncé ou clair.
Positionné sur des fronts de sciences au niveau international, le travail de l’équipe SAS intègre des approches de génétique, de phytochimie, de physiologie et d’agronomie afin de mieux comprendre les déterminants génétiques et moléculaires de l’allélopathie et ainsi développer des variétés allélopathiques chez les plantes cultivées pour la régulation biologique des adventices.
En septembre 2022, Sophie Jasinski (chargée de recherche) et Alain Lécureuil (technicien) viendront renforcer l’équipe.
Références
1 Lopez-Obando M, et al. Plant cell 4;33(11):3487-3512. https://doi.org/10.1093/plcell/koab217 (2021)
2 Bonhomme S and Guillory A, J Exp Bot https://doi.org/10.1093/jxb/erac186 (2022)
3 de Saint Germain A, et al. Front Plant Sci https://doi.org/10.3389/fpls.2022.887347 (2022)
4 Daignan Fornier S, et al, J Nat Prod https://doi.org/10.1021/acs.jnatprod.2c00282 (2022)
5 Guercio A, M, et al. Nat Com Biol https://doi.org/10.1038/s42003-022-03085-6 (2022)
Retour
Fait marquant scientifique du département INRAE BAP
Équipe "Signalisation Allélopathique et Strigolactone" SAS
Publications associées
> de Saint Germain A, Clavé G, Schouveiler p, Pillot JP , Singh A-V, Chevalier A, Suzanne Daignan Fornier, Ambre Guillory, Sandrine Bonhomme, Catherine Rameau, Boyer F-D, Expansion of the Strigolactone Profluorescent Probes Repertory: The Right Probe for the Right Application, Front Plant Sci https://doi.org/10.3389/fpls.2022.887347 (2022)
> Daignan Fornier S, de Saint Germain A, Retailleau P, Pillot J-P, Quentin Taulera Q,Andna L, L Miesch L, Rochange S, Pouvreau J-B, Boyer F-D, Noncanonical Strigolactone Analogues Highlight Selectivity for Stimulating Germination in Two Phelipanche ramosa Populations, Nat Prod https://doi.org/10.1021/acs.jnatprod.2c00282 (2022)