Rôle essentiel de la protéine « navette » SGS3 dans l’inactivation des gènes chez la plante Arabidopsis thaliana
Un crible génétique in planta a permis d’identifier les acteurs clefs des étapes cytoplasmiques du RNAi : la protéine RDR6 qui transforme les ARN aberrants en ARN double brin, les protéines DCL qui clivent les ARN double brin en siRNA, et la protéine AGO1 qui les prend en charge pour dégrader les ARNm correspondant (voir figure). Cependant, la façon dont les ARN aberrants sont sélectionnés dans le noyau et amenés à la machinerie du RNAi dans le cytoplasme restait mystérieuse. L’article publié dans Nature Communications montre que la protéine SGS3 navigue entre le noyau et le cytoplasme. Dans le noyau, elle interagit avec le facteur de remodelage de la chromatine CHR11 et se lie aux ARN aberrants tandis que dans le cytoplasme elle interagit avec l’ARN polymérase RDR6. Le séquençage des petits ARN produits lorsque la machinerie de contrôle de la qualité des ARN est perturbée par une infection virale a montré que la très grande majorité des gènes produisant ces petits ARNs fixent CHR11. Cette étude a permis d’élaborer un modèle dans lequel SGS3 prend en charge les ARN aberrants transcrits dans le noyau par les gènes qui fixent CHR11 et les exporte dans le cytoplasme où ils sont transformés en ARN double-brin par RDR6 afin d’initier la production de siRNA, soit sur une fraction importante des gènes du génome lors d’une infection virale, soit ponctuellement sur un gène précis lors de l’ajout d’une copie transgénique.
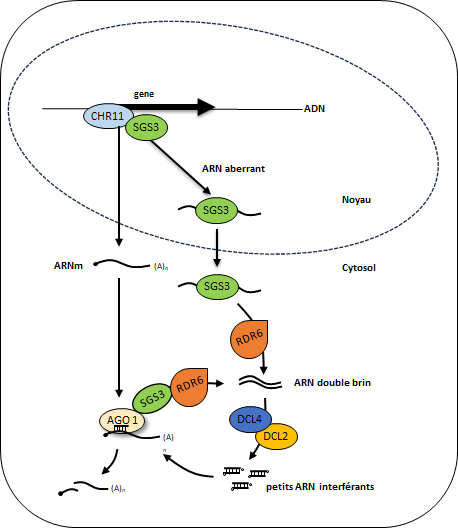
L’identification des gènes endogènes susceptibles de s’engager dans la voie du RNAi lorsque le système de contrôle qualité des ARN de la plante est perturbé permet d’alerter sur d’éventuels risques d’inactivation. Ces risques peuvent survenir lors de modifications de l’expression de ces gènes par transgénèse, voire même par modification ciblée du génome.
Une recherche développée à l’Institut Jean-Pierre Bourgin - Sciences du Végétal en collaboration.
Retour
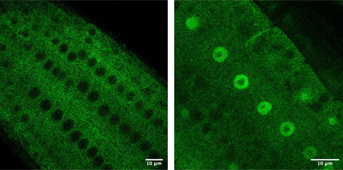
Légende :
A gauche : Localisation diffuse de SG3-GFP (marqué en vert) dans les cellules de racine d’Arabette
A droite : blocage de SGS3-GFP (marqué en vert) dans le noyau des cellules de racine d’Arabette (après traitement par un inhibiteur d’export nucléaire)
Fait marquant IJPB
Référence :
Elmayan T, Blein, T, Elvira-Matelot, E., Le Masson, I, Christ, A, Bouteiller, N, Crespi, MD, Vaucheret, H. Arabidopsis SGS3 is recruited to chromatin by CHR11 to select RNA that initiate siRNA production. Nature Communications 2025.
doi : https://doi.org/10.1038/s41467-025-57394-5
Contact : Hervé Vaucheret, contact
Équipe IJPB :
équipe "Epigénétique et petits ARNs" epiARN
Équipe IPS2 collaboratrice :
"Les ARN non-codants : des acteurs de la plasticité développementale de la racine" REGARN