La diversité des séquences cis-régulatrices d’Arabidopsis thaliana et du maïs révélée par une cartographie des régions 5’ et 3’ proximales des gènes
Avec le développement du séquençage à haut débit, la séquence complète de nombreux génomes est aujourd’hui disponible. Néanmoins, un des défis majeurs de la génomique est de décoder ces séquences brutes en informations utiles. Dans ce contexte, un des leviers potentiels est la recherche d’éléments régulateurs en cis qui sont de courtes séquences d’ADN contrôlant l’expression des gènes1. Ces séquences sont présentes en forte densité dans les régions proximales des gènes entourant en 5’ le site d’initiation de la transcription et en 3’ le site de terminaison de la transcription. La caractérisation de ces séquences cis-régulatrices proximales reste encore lacunaire, bien que des travaux expérimentaux et computationnels aient permis de faire progresser nos connaissances.
Grâce au financement du projet PLMViewer (2020-2023) par Plant2Pro, une méthode d’analyse de novo basée sur l’hypothèse de contraintes topologiques de la position des séquences cis-régulatrices par rapport au site d’initiation ou de terminaison de la transcription, a été mise en œuvre. Elle a permis de fournir une carte des séquences d’ADN préférentiellement localisées (PLM) d’Arabidopsis thaliana et du maïs mettant en évidence la structure et la fonction des séquences cis-régulatrices proximales chez ces deux espèces végétales. Trois groupes de PLM ont ainsi été identifiés dans chacune des régions étudiées. Les comparaisons réalisées ont aussi révélé que des PLM sont partagés entre les deux espèces végétales, notamment dans la région 3’-proximale. Par ailleurs, 79% des PLM identifiés n’ont pu être assignés à des sites de fixation de facteurs de transcription (tPLM) ou de microARN (miPLM). Ces séquences non assignées (uPLM) apportent des prédictions fonctionnelles distinctes de celles fournies par les tPLM et les miPLM. De plus, une partie d’entres elles a été appuyée par des données expérimentales de type MOA-seq, suggérant que les uPLM correspondraient à de nouvelles séquences cis-régulatrices proximales.
Cette étude illustre la richesse et la diversité des PLM présents dans les régions proximales des gènes d’Arabidopsis thaliana et du maïs, contribuant non seulement aux interactions PLM-facteur de transcription, mais aussi aux interactions PLM-microARN et PLM-protéine de liaison à l’ARN2. L’analyse détaillée des PLM chez d’autres espèces de plantes à fleurs est prometteuse pour améliorer le pouvoir prédictif des modèles de sélection.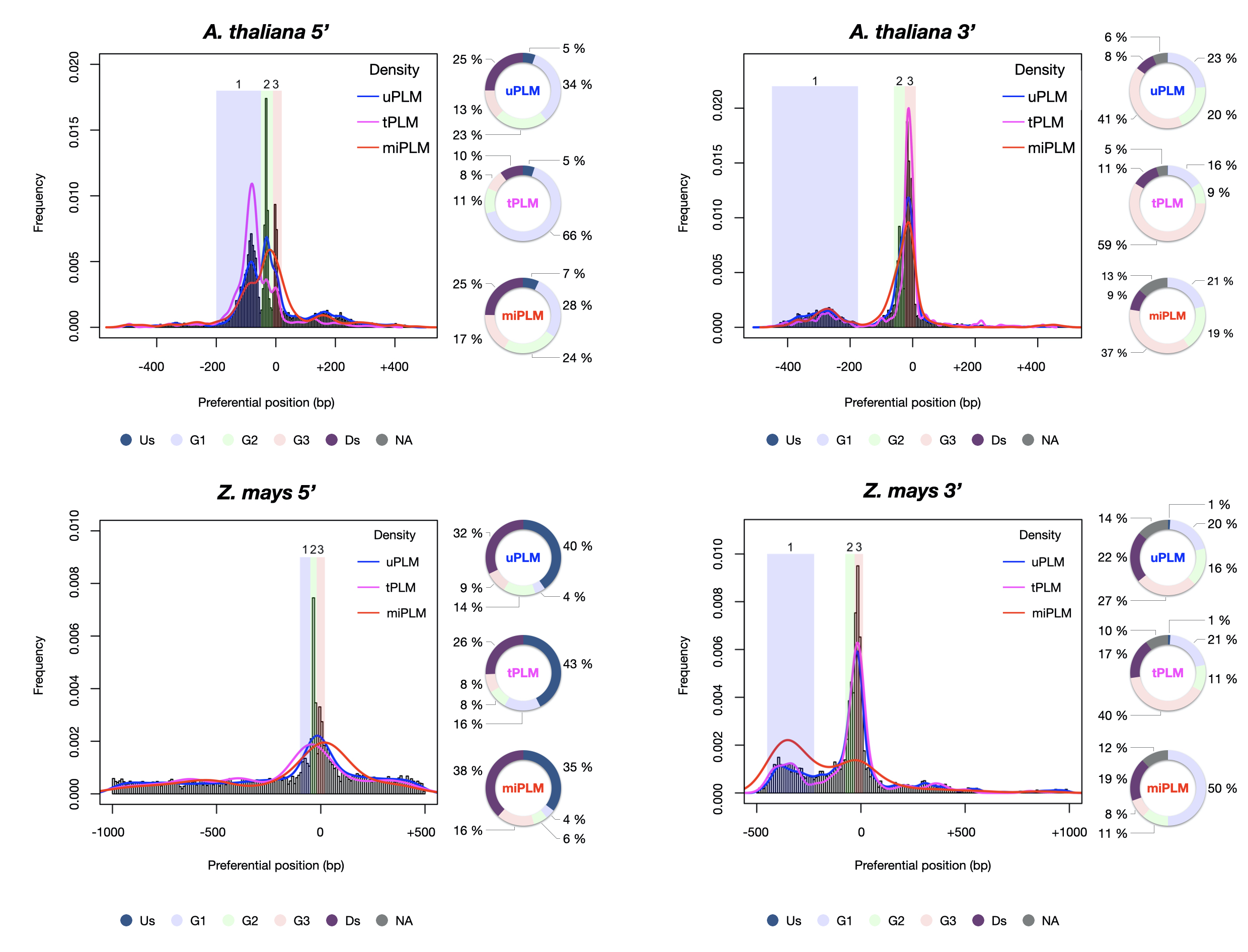
Légende : Diversité des PLM présents dans les régions 5’ et 3’proximales des gènes d’Arabidopsis thaliana et du maïs. uPLM : PLM non assigné ; tPLM : site de fixation de facteur de transcription ; miPLM : séquences présentant des homologies avec des microARN ; Us : région située en amont des trois groupes identifiés dans chacune des régions proximales ; G1, G2 et G3 : groupes 1, 2 et 3 ; Ds : région située en aval des trois groupes ; NA : région située entre les groupes lorsqu’ils ne sont pas juxtaposés.
Références bibliographiques
1Schmitz JS, Grotewold E, Stam M (2021). Cis-regulatory sequences in plants: Their importance, discovery, and future challenges. The Plant Cell 34(2): 718–741 https://doi.org/10.1093/plcell/koab281
2Rozière J, Guichard C, Brunaud V, Martin ML, Coursol S (2022). A comprehensive map of preferentially located motifs reveals distinct proximal cis-regulatory sequences in plants. Front. Plant Sci. 12 October 2022 Sec. Plant Bioinformatics https://doi.org/10.3389/fpls.2022.976371
Retour

zoom
Fait marquant IJPB du département BAP
Équipe "Qualité de la biomasse et interactions avec la sécheresse" QUALIBIOSEC
Publication associée
Rozière J, Guichard C, Brunaud V, Martin ML, Coursol S (2022).
A comprehensive map of preferentially located motifs reveals distinct proximal cis-regulatory sequences in plants. Front. Plant Sci. 12 October 2022 Sec. Plant Bioinformatics https://doi.org/10.3389/fpls.2022.976371