Quantification semi-automatique des signaux de phosphorylation d’une histone impliquée dans la division chez Arabidopsis
Biologie cellulaire
Analyse d'images 3D
modification post-traductionnelle
Nouvel outil d’analyse d’images de microscopie 3D : une publication des équipes SPACE et MIN dans New Phytologist
De nombreux aspects de la division cellulaire sont contrôlés par des modifications post-traductionnelles des histones. Parmi elles, la phosphorylation de la sérine 10 de l'histone H3 est cruciale pour la ségrégation correcte des chromosomes pendant la mitose. L'objectif de cette étude était de mesurer avec précision la dynamique de cette phosphorylation pendant la mitose dans les cellules végétales afin de mieux comprendre les voies moléculaires impliquées.
Un pipeline d'analyse d'images 3D obtenues après immunolocalisation de l’H3S10ph a été développé pour permettre la quantification semi-automatisée de la phosphorylation de H3S10 dans les cellules mitotiques des racines d'Arabidopsis. De plus, une nouvelle méthode a été conçue pour corriger l'atténuation du signal dans la profondeur de l'image, phénomène inhérent à ce type d'imagerie 3D. Cette méthode repose sur la mesure directe de l'intensité des objets d'intérêt et la caractérisation de leur atténuation dans la profondeur de l'image. En tenant compte du profil d'atténuation du signal dans la pile d'images, cette correction améliore considérablement la précision et la robustesse statistique des analyses. Grâce à ce pipeline, des différences subtiles dans les niveaux de H3S10ph ont été détectées entre des mutants présentant une activité phosphatase PP2A altérée, suggérant une implication indirecte des enzymes PP2A dans la régulation des niveaux de H3S10ph.
Cet outil offre de nouvelles possibilités pour étudier ces voies de régulation chez les plantes en tirant parti des vastes ressources génétiques disponibles chez Arabidopsis. Il peut également être adapté pour étudier d'autres modifications post-traductionnelles des histones ou, plus généralement, tout signal 3D discret.
Une recherche développée à l’Institut Jean-Pierre Bourgin - Sciences du Végétal en collaboration.
Un pipeline d'analyse d'images 3D obtenues après immunolocalisation de l’H3S10ph a été développé pour permettre la quantification semi-automatisée de la phosphorylation de H3S10 dans les cellules mitotiques des racines d'Arabidopsis. De plus, une nouvelle méthode a été conçue pour corriger l'atténuation du signal dans la profondeur de l'image, phénomène inhérent à ce type d'imagerie 3D. Cette méthode repose sur la mesure directe de l'intensité des objets d'intérêt et la caractérisation de leur atténuation dans la profondeur de l'image. En tenant compte du profil d'atténuation du signal dans la pile d'images, cette correction améliore considérablement la précision et la robustesse statistique des analyses. Grâce à ce pipeline, des différences subtiles dans les niveaux de H3S10ph ont été détectées entre des mutants présentant une activité phosphatase PP2A altérée, suggérant une implication indirecte des enzymes PP2A dans la régulation des niveaux de H3S10ph.
Cet outil offre de nouvelles possibilités pour étudier ces voies de régulation chez les plantes en tirant parti des vastes ressources génétiques disponibles chez Arabidopsis. Il peut également être adapté pour étudier d'autres modifications post-traductionnelles des histones ou, plus généralement, tout signal 3D discret.
Une recherche développée à l’Institut Jean-Pierre Bourgin - Sciences du Végétal en collaboration.
Retour
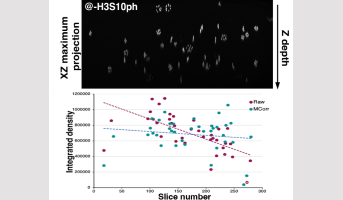
Légende : En haut, projection en XZ d’une pile d'images 3D de racine, après immunolocalisation, montrant la diminution de l’intensité des signaux H3S10 selon la profondeur (Z). En bas, graphe montrant la correction de l'atténuation de ce signal grâce au pipeline développé dans cette étude (différence de la pente entre MCorr et Raw data)
Fait marquant IJPB
Référence
Kelemen A, Uyttewaal M, Máthé C, Andrey P, Bouchez D, Pastuglia M. Semiautomatic quantification of 3D Histone H3 phosphorylation signals during cell division in Arabidopsis root meristems. New Phytol 2025. doi: 10.1111/nph.70365.
Contact : Martine Pastuglia
Équipes IJPB
> Contrôle spatial de la division cellulaire SPACE
> Modélisation et Imagerie Numérique MiN
Equipe collaboratrice
Department of Botany, Faculty of Science and Technology, University of Debrecen, Debrecen, Hongrie
Fait marquant IJPB
Référence
Kelemen A, Uyttewaal M, Máthé C, Andrey P, Bouchez D, Pastuglia M. Semiautomatic quantification of 3D Histone H3 phosphorylation signals during cell division in Arabidopsis root meristems. New Phytol 2025. doi: 10.1111/nph.70365.
Contact : Martine Pastuglia
Équipes IJPB
> Contrôle spatial de la division cellulaire SPACE
> Modélisation et Imagerie Numérique MiN
Equipe collaboratrice
Department of Botany, Faculty of Science and Technology, University of Debrecen, Debrecen, Hongrie