Nos recherches visent à concevoir des approches originales en analyse d’images, statistiques spatiales et modélisation computationnelle pour mieux comprendre le développement et le fonctionnement des plantes.
*** Une offre de post-doc disponible, une autre à ouvrir bientôt (voir Emploi) ***
Motivation
Une question centrale en biologie du développement est de comprendre comment différents facteurs, processus et échelles sont intégrés au cours de la morphogenèse pour produire des patrons d’organisation et des formes spécifiques au niveau des tissus et des organes. Nos recherches ont pour objectif de développer de nouvelles méthodes pour visualiser, quantifier et modéliser les données fournies par les images de microscopie afin de répondre à ces questionnements.
Thématiques
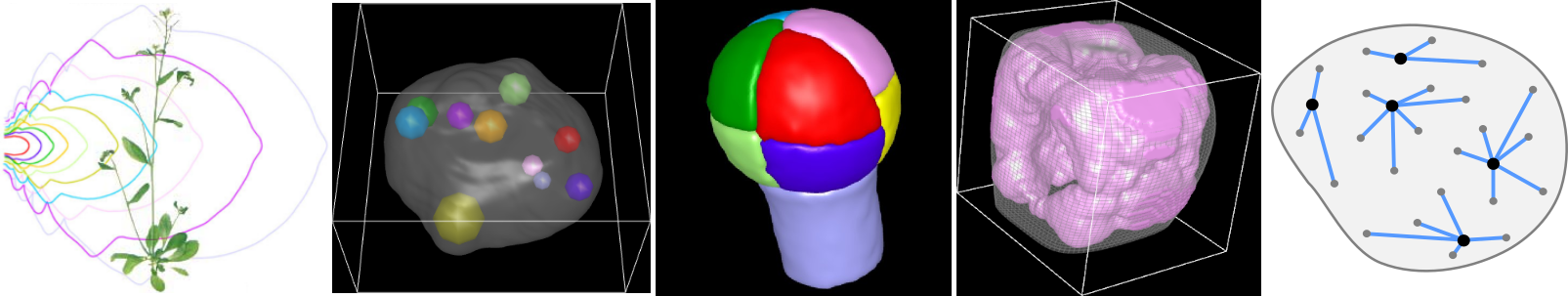
Nos recherches sont structurées autour de trois axes :
- traitement et analyse des images : nous développons des stratégies algorithmiques pour extraire les informations pertinentes des images et construire des représentations statistiques à partir d’ensembles d’observations individuelles ;
- statistiques spatiales : nous développons des méthodes pour cartographier de façon statistique et en trois dimensions des organisations spatiales au sein des systèmes biologiques et pour déchiffrer les principes architecturaux qui les sous-tendent ;
- modélisation computationnelle : nous développons des modèles informatiques prédictifs pour comprendre les mécanismes responsables de la mise en place et de la régulation des patrons d’organisation et de la forme des organes végétaux.
Motivation
Une question centrale en biologie du développement est de comprendre comment différents facteurs, processus et échelles sont intégrés au cours de la morphogenèse pour produire des patrons d’organisation et des formes spécifiques au niveau des tissus et des organes. Nos recherches ont pour objectif de développer de nouvelles méthodes pour visualiser, quantifier et modéliser les données fournies par les images de microscopie afin de répondre à ces questionnements.
Thématiques
Nos recherches sont structurées autour de trois axes :
- traitement et analyse des images : nous développons des stratégies algorithmiques pour extraire les informations pertinentes des images et construire des représentations statistiques à partir d’ensembles d’observations individuelles ;
- statistiques spatiales : nous développons des méthodes pour cartographier de façon statistique et en trois dimensions des organisations spatiales au sein des systèmes biologiques et pour déchiffrer les principes architecturaux qui les sous-tendent ;
- modélisation computationnelle : nous développons des modèles informatiques prédictifs pour comprendre les mécanismes responsables de la mise en place et de la régulation des patrons d’organisation et de la forme des organes végétaux.
Publications choisies (voir onglet "Publications" pour la liste complète)
Laruelle E, Belcram K, Trubuil A, Palauqui J-C and Andrey P (2022). Large-scale analysis and computer modeling reveal hidden regularities behind variability of cell division patterns in Arabidopsis thaliana embryogenesis. eLife, 11, e79224. https://doi.org/10.7554/eLife.79224.
Arpòn J, Sakai K, Gaudin V and Andrey P (2021). Spatial modeling of biological patterns shows multiscale organization of Arabidopsis thaliana heterochromatin. Scientific Reports, 11, 323. https://doi.org/10.1038/s41598-020-79158-5.
Burguet J and Andrey P (2020). Edge correction for intensity estimation of 3D heterogeneous point processes from replicated data. Spatial Statistics, 36, 100421. https://doi.org/10.1016/j.spasta.2020.100421.
Moukhtar J, Trubuil A, Belcram K, Legland D, Khadir Z, Urbain A, Palauqui J-C and Andrey P (2019). Cell geometry determines symmetric and asymmetric division plane selection in Arabidopsis early embryos. PLoS Computational Biology, 15, e1006771. https://doi.org/10.1371/journal.pcbi.1006771.
Arganda-Carreras I and Andrey P (2017). Designing Image Analysis Pipelines in Light Microscopy: A Rational Approach. Methods in Molecular Biology, 1563, 185-207. https://doi.org/10.1007/978-1-4939-6810-7_13.
Biot E, Cortizo M, Burguet J, Kiss A, Oughou M, Maugarny-Calès A, Gonçalves B, Adroher B, Andrey P, Boudaoud A and Laufs P (2016). Multiscale quantification of morphodynamics: MorphoLeaf, software for 2-D shape analysis. Development, 143, 3417-3428. https://doi.org/10.1242/dev.134619.
Biot E, Crowell E, Burguet J, Höfte H, Vernhettes S and Andrey P (2016). Strategy and software for the statistical spatial analysis of 3D intracellular distributions. Plant Journal, 87, 230-242. https://doi.org/10.1111/tpj.13189.
Legland D, Arganda-Carreras I and Andrey P (2016). MorphoLibJ: integrated library and plugins for mathematical morphology with ImageJ. Bioinformatics, 32, 3532-3534. https://doi.org/10.1093/bioinformatics/btw413.
Burguet J and Andrey P (2014). Statistical comparison of spatial point patterns in biological imaging. PLoS ONE, 9, e87759. https://doi.org/10.1371/journal.pone.0087759.
Andrey P, Kiêu K, Kress C, Lehmann G, Tirichine L, Liu Z, Biot E, Adenot P-G, Hue-Beauvais C, Houba-Hérin N, Duranthon V, Devinoy E, Beaujean N, Gaudin V, Maurin Y and Debey P (2010). Statistical analysis of 3D images detects regular spatial distributions of centromeres and chromocenters in animal and plant nuclei. PLoS Computational Biology, 6, e1000853. https://doi.org/10.1371/journal.pcbi.1000853.
Andrey P and Maurin Y (2005). Free-D: an integrated environment for three-dimensional reconstruction from serial sections. Journal of Neuroscience Methods, 145, 233-244. https://doi.org/10.1016/j.jneumeth.2005.01.006.
Laruelle E, Belcram K, Trubuil A, Palauqui J-C and Andrey P (2022). Large-scale analysis and computer modeling reveal hidden regularities behind variability of cell division patterns in Arabidopsis thaliana embryogenesis. eLife, 11, e79224. https://doi.org/10.7554/eLife.79224.
Arpòn J, Sakai K, Gaudin V and Andrey P (2021). Spatial modeling of biological patterns shows multiscale organization of Arabidopsis thaliana heterochromatin. Scientific Reports, 11, 323. https://doi.org/10.1038/s41598-020-79158-5.
Burguet J and Andrey P (2020). Edge correction for intensity estimation of 3D heterogeneous point processes from replicated data. Spatial Statistics, 36, 100421. https://doi.org/10.1016/j.spasta.2020.100421.
Moukhtar J, Trubuil A, Belcram K, Legland D, Khadir Z, Urbain A, Palauqui J-C and Andrey P (2019). Cell geometry determines symmetric and asymmetric division plane selection in Arabidopsis early embryos. PLoS Computational Biology, 15, e1006771. https://doi.org/10.1371/journal.pcbi.1006771.
Arganda-Carreras I and Andrey P (2017). Designing Image Analysis Pipelines in Light Microscopy: A Rational Approach. Methods in Molecular Biology, 1563, 185-207. https://doi.org/10.1007/978-1-4939-6810-7_13.
Biot E, Cortizo M, Burguet J, Kiss A, Oughou M, Maugarny-Calès A, Gonçalves B, Adroher B, Andrey P, Boudaoud A and Laufs P (2016). Multiscale quantification of morphodynamics: MorphoLeaf, software for 2-D shape analysis. Development, 143, 3417-3428. https://doi.org/10.1242/dev.134619.
Biot E, Crowell E, Burguet J, Höfte H, Vernhettes S and Andrey P (2016). Strategy and software for the statistical spatial analysis of 3D intracellular distributions. Plant Journal, 87, 230-242. https://doi.org/10.1111/tpj.13189.
Legland D, Arganda-Carreras I and Andrey P (2016). MorphoLibJ: integrated library and plugins for mathematical morphology with ImageJ. Bioinformatics, 32, 3532-3534. https://doi.org/10.1093/bioinformatics/btw413.
Burguet J and Andrey P (2014). Statistical comparison of spatial point patterns in biological imaging. PLoS ONE, 9, e87759. https://doi.org/10.1371/journal.pone.0087759.
Andrey P, Kiêu K, Kress C, Lehmann G, Tirichine L, Liu Z, Biot E, Adenot P-G, Hue-Beauvais C, Houba-Hérin N, Duranthon V, Devinoy E, Beaujean N, Gaudin V, Maurin Y and Debey P (2010). Statistical analysis of 3D images detects regular spatial distributions of centromeres and chromocenters in animal and plant nuclei. PLoS Computational Biology, 6, e1000853. https://doi.org/10.1371/journal.pcbi.1000853.
Andrey P and Maurin Y (2005). Free-D: an integrated environment for three-dimensional reconstruction from serial sections. Journal of Neuroscience Methods, 145, 233-244. https://doi.org/10.1016/j.jneumeth.2005.01.006.

Responsable :
Philippe Andrey