 |
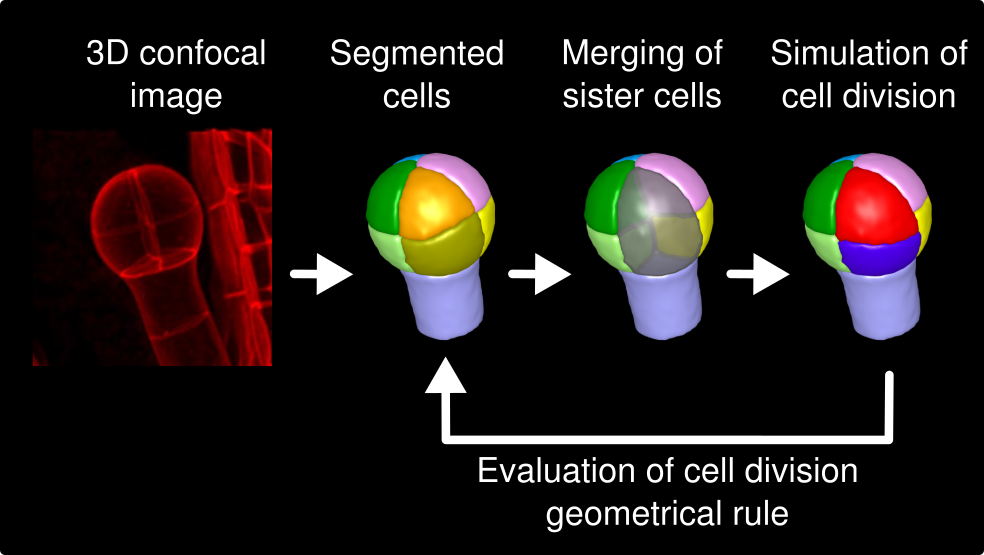
Modélisation des patrons de division dans l’embryon d’Arabidopsis (Moukhtar et al 2019, Laruelle et al 2022) : nous avons développé un nouveau modèle de division cellulaire 3D orienté-image. Avec ce modèle, nous avons montré qu’une règle géométrique unique pouvait expliquer les patrons de divisions 3D dans l’embryon précoce d’Arabidopsis. |
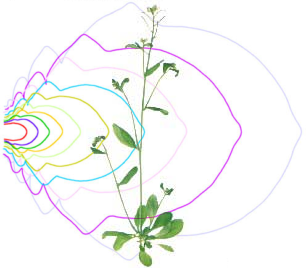 |
Quantification de dynamiques morphogénétiques (Biot et al 2016, Oughou et al 2023) : nous avons développé un pipeline et un outil logiciel permettant de reconstituer la dynamique de croissance d’organes à partir d’images d’échantillons à différents stades de développement. |
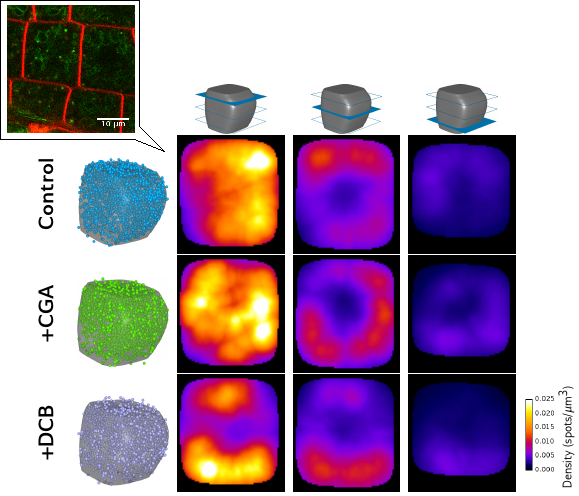 |
Modélisation statistique de distributions intra-cellulaires (Biot et al 2016b) : nous avons développé un pipeline et un outil logiciel pour inférer, à partir de collections d’images 3D, la répartition statistique de compartiments intra-cellulaires au sein d’une cellule moyenne. |
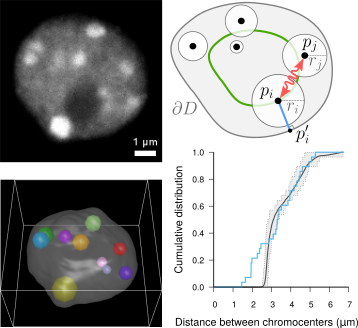 |
Modélisation statistique d’interactions spatiales (Andrey et al 2010, Arpon et al 2018, Arpon et al 2021) : nous développons une approche de modélisation spatiale pour déchiffrer les règles qui régissent la répartition d’objets biologiques dans l’espace. Avec cette approche, nous avons montré que, dans les noyaux de cellules de feuilles d’Arabidopsis, les chromocentres suivaient une organisation multi-échelle complexe. |
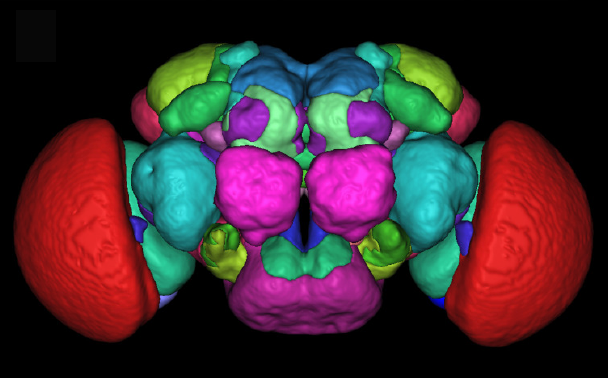 |
Atlasing statistique de données d'imagerie (Arganda-Carreras et al 2018) : nous avons exploité une collection d’images annotées de cerveaux de Drosophile adulte pour élaborer un atlas statistique 3D du cerveau et de ses principales structures. Cet atlas permet la normalisation spatiale et la comparaison d’images de patrons d’expression de gènes. |

Responsable :
Philippe Andrey