1) Dynamique des Corps Lipidiques : Sabine d’Andréa, Isabelle Bouchez, Carine Deruyffelaere.
Dans les graines sèches, les réserves lipidiques stockées dans les CL restent stables plusieurs années grâce aux protéines majeures qui les entourent, les oléosines. Lorsque ces graines germent, elles mobilisent leurs lipides de réserve pour permettre la croissance de la plantule. Nous avons voulu comprendre comment les CL, remarquablement stables dans la graine sèche, pouvaient être rapidement dégradés lors de la germination. Nous avons montré que la dégradation des oléosines précède la dégradation des lipides de réserve. Les oléosines sont étiquetées pour la dégradation par des modifications appelées ubiquitinations, puis extraites des CL pour être ensuite dégradées par protéolyse. Nous avons ensuite voulu comprendre comment des protéines aussi ancrées aux CL que les oléosines pouvaient être extraites de la surface des CL. Nous avons ainsi découvert qu’une protéine appelée PUX10 est nécessaire pour extraire correctement les oléosines ubiquitinées de la surface des CL chez les plantules d’Arabidopsis. PUX10 est localisée sur les CL et se lie aux oléosines ubiquitinées. Elle interagit également avec une ATPase nommée CDC48A, connue pour extraire de manière sélective les protéines mal repliées du réticulum endoplasmique. PUX10 est ainsi une protéine adaptatrice qui recrute cette ATPase sur les CL favorisant de cette manière l'extraction des oléosines ubiquitinées avant leur dégradation protéolytique. Nous proposons que PUX10 et CDC48A soient au cœur d'une nouvelle machinerie de dégradation associée aux CL, et que nous avons nommé LDAD (Lipid Droplet-Associated Degradation) (Deruyffelaere 2018, 2015).
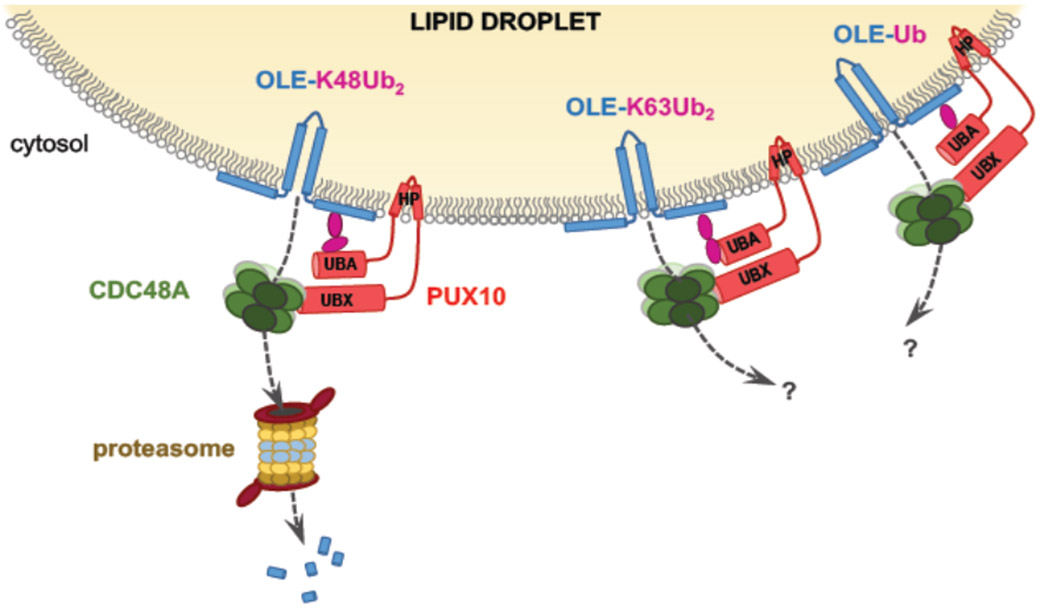
Nos résultats suggèrent aussi fortement l’existence de sous populations de CL.

Nos résultats suggèrent aussi fortement l’existence de sous populations de CL.
2) Topologie et accessibilité relative des protéines associées aux CL de plantes (Christelle Louis-Mondésir, Thierry Chardot, Yann Gohon).
Les oléosines sont des protéines très peu solubles en milieu aqueux, et elles sont insérées à la surface des CL dans une hémi membrane de phospholipides, ce qui est exceptionnel dans la nature. L’étude de leur structure ne peut être réalisée qu’en présence de surfactants pour les maintenir solubles, ou bien in situ dans leur environnement naturel, les CL.
Notre partenariat étroit avec le synchrotron SOLEIL (ligne de lumière DISCO, St. Aubin) a permis, à l'aide du dichroïsme circulaire par rayonnement synchrotron (SRCD), d’étudier le repliement de l’oléosine majoritaire (S3) des CL de graines dans son environnement naturel. Au sein des CL, l’oléosine S3 est repliée majoritairement et de manière originale avec des brins béta. Afin de délimiter avec précision comment cette protéine était insérée à la surface des CL, nous avons développé une approche de protéomique structurale avec la plateforme de protéomique PAPSSO (Le Moulon) et la ligne de lumière Métrologie du Synchrotron SOLEIL. Le rayonnement synchrotron a permis de produire par photolyse de l’eau des radicaux hydroxyle (•OH). Les •OH ont une durée de vie très courte (~ μs) et permettent d’oxyder les résidus d'acides aminés accessibles à l'eau. La protéomique a permis d’identifier dans des CL purifiés et à l’échelle des acides aminés les régions de la protéine oxydée, en contact avec l’eau. Les prolines ne sont pas oxydées et se trouvent donc dans le cœur de triglycérides des CL.
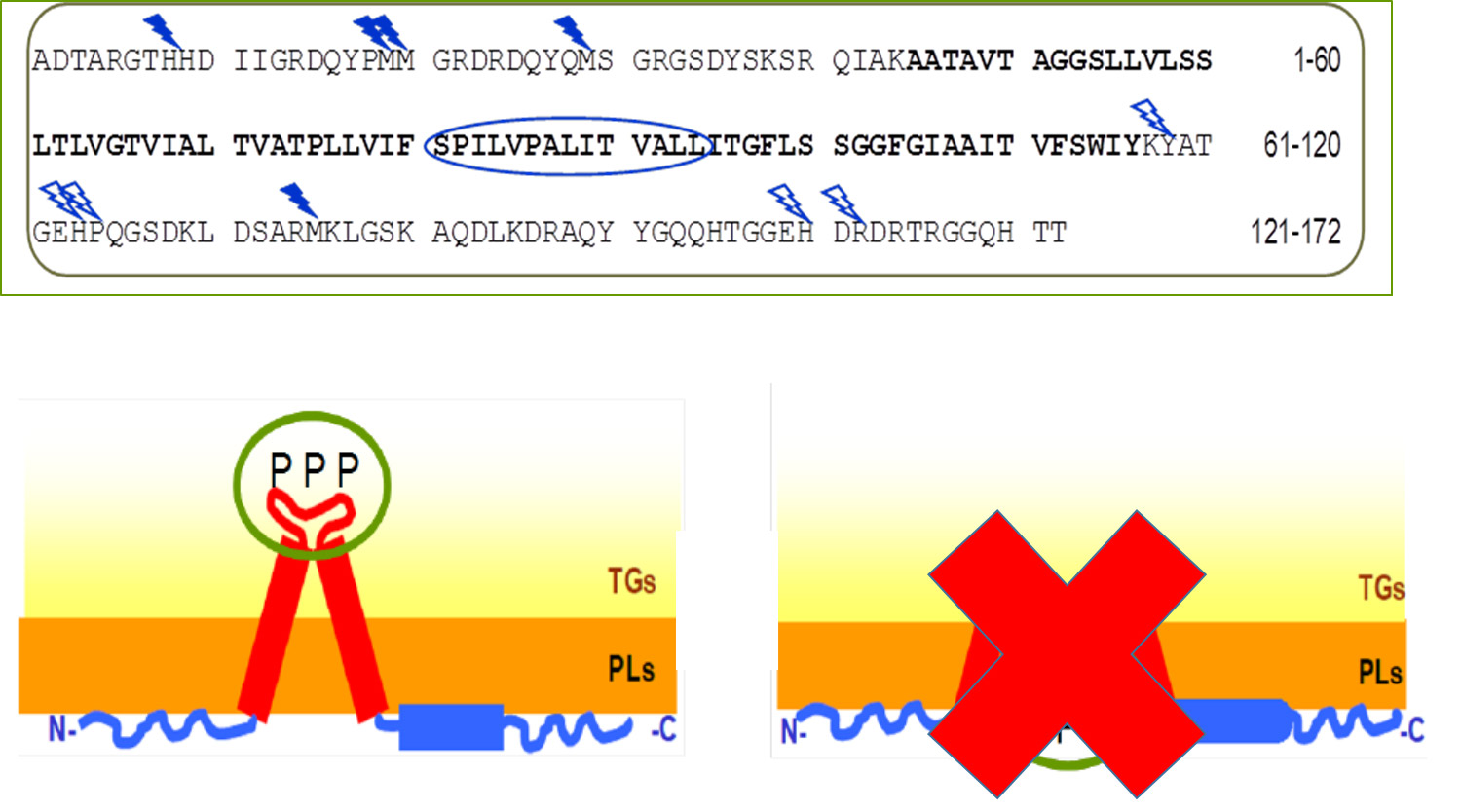
Cette première étude de protéomique structurale sur organites intacts a permis de déterminer la topologie de l'oléosine S3 dans son environnement naturel (Baud 2017, Jolivet 2017).
Notre partenariat étroit avec le synchrotron SOLEIL (ligne de lumière DISCO, St. Aubin) a permis, à l'aide du dichroïsme circulaire par rayonnement synchrotron (SRCD), d’étudier le repliement de l’oléosine majoritaire (S3) des CL de graines dans son environnement naturel. Au sein des CL, l’oléosine S3 est repliée majoritairement et de manière originale avec des brins béta. Afin de délimiter avec précision comment cette protéine était insérée à la surface des CL, nous avons développé une approche de protéomique structurale avec la plateforme de protéomique PAPSSO (Le Moulon) et la ligne de lumière Métrologie du Synchrotron SOLEIL. Le rayonnement synchrotron a permis de produire par photolyse de l’eau des radicaux hydroxyle (•OH). Les •OH ont une durée de vie très courte (~ μs) et permettent d’oxyder les résidus d'acides aminés accessibles à l'eau. La protéomique a permis d’identifier dans des CL purifiés et à l’échelle des acides aminés les régions de la protéine oxydée, en contact avec l’eau. Les prolines ne sont pas oxydées et se trouvent donc dans le cœur de triglycérides des CL.

Cette première étude de protéomique structurale sur organites intacts a permis de déterminer la topologie de l'oléosine S3 dans son environnement naturel (Baud 2017, Jolivet 2017).
3) Informations fonctionnelles et structurales sur le fonctionnement des DGAT de différentes familles (Frank Jagic, Christelle LouisMondésir, Sihem Hentati, Justine Tisserand, Pierre Briozzo, Thierry Chardot)
La voie de Kennedy permet le stockage stable des acides gras chez les eucaryotes sous forme de TG, appelés huiles chez les plantes. Trois acyltransférases membranaires catalysent l’addition stéréospécifique et successive d’acides gras sur un squelette glycérol. Les DGAT (diacylglycérol acyltransférases) ajoutent l’acide gras final en position sn-3. Plus de 130 acides gras peuvent incorporés dans les huiles végétales, ce qui leur confère des propriétés physiques, chimiques et nutritionnelles particulières. Du fait de leur impact sur le rendement et la qualité de l'huile, les DGAT sont des cibles de choix pour l'ingénierie des huiles. Elles appartiennent à 3 familles phylogénétiques distinctes. Les deux premières, majoritairement exprimées dans les graines au niveau du réticulum endoplasmique, sont impliquées dans l’accumulation de l’huile.
Les DGAT3 ont été identifiées pour la première fois en 2006. Leur localisation dans le cytosol suggère que ces enzymes devraient être solubles, et faire partie d'une voie alternative pour la synthèse de l’huile. La DGAT3 est donc une cible originale pour les études métaboliques, enzymatiques et structurales.
Un examen attentif de la séquence protéique a montré une région C-terminale de type ferrédoxine conservée dans les DGAT3 de plantes. Cette région est potentiellement associée à un cluster 2Fe-2S. La protéine recombinante purifiée AtDGAT3 avait une couleur brun-rouge, s'estompant avec le temps. Grâce au laboratoire du professeur Fontecave (Collège de France, Paris), nous avons purifié cette protéine sans oxygène et démontré, en utilisant la résonance paramagnétique électromagnétique, la présence un cluster [2Fe-2S].
![AtDGAT3 possède un centre [2Fe-2S]](https://ijpb.versailles.inrae.fr/uploads/2020/03/20/dyscol-projets-3-tif.jpg)
C'est la première fois que l’on démontre qu’une DGAT possède un centre [Fe-S] (Aymé 2018).
Les DGAT3 ont été identifiées pour la première fois en 2006. Leur localisation dans le cytosol suggère que ces enzymes devraient être solubles, et faire partie d'une voie alternative pour la synthèse de l’huile. La DGAT3 est donc une cible originale pour les études métaboliques, enzymatiques et structurales.
Un examen attentif de la séquence protéique a montré une région C-terminale de type ferrédoxine conservée dans les DGAT3 de plantes. Cette région est potentiellement associée à un cluster 2Fe-2S. La protéine recombinante purifiée AtDGAT3 avait une couleur brun-rouge, s'estompant avec le temps. Grâce au laboratoire du professeur Fontecave (Collège de France, Paris), nous avons purifié cette protéine sans oxygène et démontré, en utilisant la résonance paramagnétique électromagnétique, la présence un cluster [2Fe-2S].
![AtDGAT3 possède un centre [2Fe-2S]](https://ijpb.versailles.inrae.fr/uploads/2020/03/20/dyscol-projets-3-tif.jpg)
C'est la première fois que l’on démontre qu’une DGAT possède un centre [Fe-S] (Aymé 2018).
Les projets 1 et 3 ont été supportés par le programme Procope, Campus France “Futuroil” 2018-2019, coordonné par T Chardot et I Feussner (Goettingen)
4) Biologie structurale et ingénierie d’enzymes : F Jagic, D Machover (Hôpital Paul Brousse), P Briozzo
L’équipe met ses compétences en matière de biologie structurale et d’ingénierie d’enzymes au service du collectif. Les principales enzymes étudiées sont des protéases à cystéine, des cytokinines oxydase, aldéhydes déshydrogénases, acyltransférases végétales, et des méthionine gamma lyase (MGL) végétales et microbiennes. Au cours des 5 dernières années l’équipe a déposé 10 structures 3D originales de protéines dans la « Protein Data Bank »
4) Biologie structurale et ingénierie d’enzymes : F Jagic, D Machover (Hôpital Paul Brousse), P Briozzo
L’équipe met ses compétences en matière de biologie structurale et d’ingénierie d’enzymes au service du collectif. Les principales enzymes étudiées sont des protéases à cystéine, des cytokinines oxydase, aldéhydes déshydrogénases, acyltransférases végétales, et des méthionine gamma lyase (MGL) végétales et microbiennes. Au cours des 5 dernières années l’équipe a déposé 10 structures 3D originales de protéines dans la « Protein Data Bank »
Responsable :
Thierry Chardot